Mi experiencia investigadora empezó durante mis estudios de biología en la Universidad de Málaga. Allí, durante los dos últimos años de carrera me incorporé al Departamento de Biología celular, Genética y Fisiología animal, estudiando el papel del receptor de ácido lisofosfatídico, LAPR1, en el control de la neurogénesis en el cerebro adulto de ratón. Esta experiencia reafirmó aún más mi vocación por la investigación y el trabajo de laboratorio.
Al finalizar mis estudios, me incorporé al laboratorio de Paola Bovolenta (Madrid), gracias a una beca de introducción de la investigación del CSIC y, posteriormente, a un contrato predoctoral de la Comunidad de Madrid. En su laboratorio me apasioné por el estudio del desarrollo del cerebro en estadios embrionarios tempranos. Me fascinó el hecho de que las distintas estructuras de nuestro cerebro (como la corteza cerebral o los ojos) derivan de pequeños grupos de células que adquieren su identidad mucho antes de que las estructuras que van a generar puedan siquiera observarse en el embrión. En el caso del cerebro, esta identidad se establece cuando las células que van a formarlo constituyen todavía una simple monocapa. Por tanto, toda la información necesaria para formar las distintas regiones del cerebro debe codificarse inicialmente en dos dimensiones. Esto es posible porque, en esa estructura en 2D (llamada placa neural), hay genes que se activan específicamente en un grupo de células y no en otras, formando un código combinatorio que define cada región de la placa neural, determinando así su “destino final”.
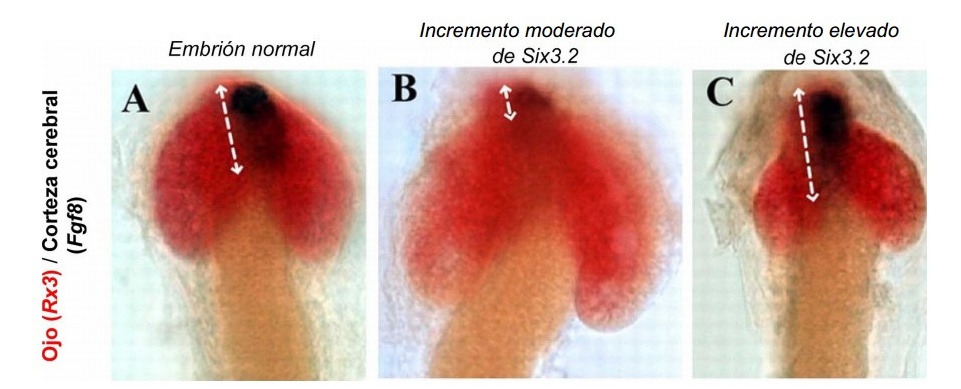
Para comprender la lógica de este proceso, estudié las funciones y la regulación de uno de esos genes (el gen Six3) demostrando que sus niveles de actividad impactan de forma importante en el desarrollo de las distintas estructuras cerebrales. Por ejemplo, una baja actividad de Six3 favorece la formación de ojo a expensas de la corteza y otras estructuras cerebrales, mientras altos niveles del mismo hacen lo contrario (Figura 1).
Otro aspecto que me fascinó fue cómo la activación precisa de los genes de desarrollo está controlada por secuencias de ADN que no codifican ningún gen (consideradas por ello durante mucho tiempo como ADN “basura”), pero que contienen la información para que un determinado gen se active de forma correcta. Para actuar, estas secuencias reguladoras (a los cuales nos referimos genéricamente como “enhancers”) necesitan establecer contacto con su gen diana mediante la formación de bucles en el ADN. Por tanto, la organización 3D del genoma juega un papel importante en la regulación génica. Este concepto comenzó a delinearse más claramente coincidiendo con los años finales de mi tesis doctoral gracias al establecimiento de nuevas técnicas para estudiar la arquitectura de nuestro genoma.
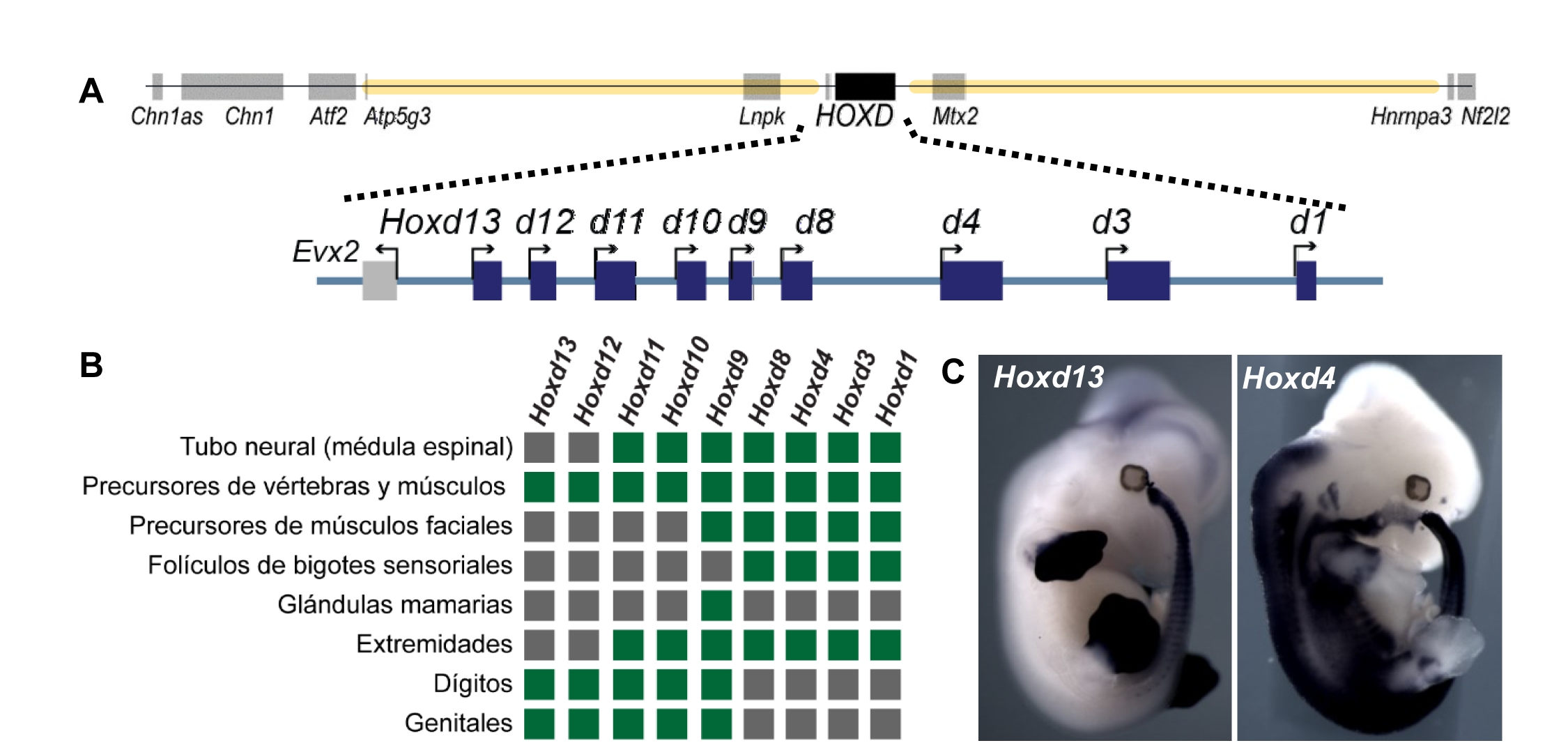
El interés por comprender estos mecanismos me llevó a unirme al laboratorio de Denis Duboule (Ginebra), donde estudié cómo la organización 3D del genoma y la actividad de los enhancers controlan la activación de los genes de desarrollo. Para ello, estudié un grupo de 9 genes, llamados genes Hoxd, que reciben al apelativo de genes arquitecto por su papel esencial en el desarrollo de distintas estructuras de nuestro cuerpo. Mis estudios demostraron que la organización 3D de la región genómica que contiene dichos genes juega un papel esencial en su regulación, permitiendo que diferentes grupos de enhancers activen combinaciones específicas de los mismos en distintas estructuras embrionarias. Además, comparando distintas especies de vertebrados, concluimos que la regulación de estos genes ha evolucionado mediante la adquisición de nuevas secuencias reguladoras, pero manteniendo una misma organización 3D del ADN (Figura 2). Durante estos años también implementé en el laboratorio el cultivo de gastruloides: agregados 3D de células que reproducen los eventos de especificación y organización celular que ocurren durante el desarrollo temprano del embrión. Esta experiencia me convenció de que este tipo de cultivos constituyen un excelente modelo para el estudio de los procesos de regulación génica ya que permiten combinar técnicas de edición genética y de secuenciación masiva, permitiendo además investigar directamente la regulación de genes humanos sin recurrir al uso de modelos experimentales de otras especies.
Después de esta experiencia, me propuse combinar los intereses científicos que caracterizaron mis etapas pre y postdoctoral para dedicarme al estudio de los mecanismos de regulación génica que operan durante el desarrollo del cerebro. En particular, decidí enfocarme en el estudio del desarrollo de la corteza cerebral humana. Para ello, me incorporé en 2020 al Instituto NeuroMyoGène (Lion, Francia) donde empecé a establear las bases metodológicas para esta investigación. En mi próxima etapa, gracias al programa Ramón y Cajal, voy a continuar desarrollando mi plan científico en España, incorporándome a un centro de gran prestigio como el Centro de Biología Molecular Severo Ochoa (CBMSO), donde dispondré de todas las infraestructuras necesarias para llevar a cabo mi investigación. Además, estaré en un entorno de excelencia investigadora como es el del campus de la Universidad Autónoma de Madrid y, más en general, el de la Comunidad de Madrid, con un gran número de centros de Investigación y laboratorios de excelencia en investigación básica y clínica.
Mi investigación se centrará en la caracterización de las secuencias reguladoras que controlan genes esenciales para el desarrollo cortical (¿cuáles son estas secuencias? ¿en qué tipos celulares actúan? ¿son activadoras o represoras?) y en el estudio de cómo la organización 3D del genoma permite el control de estas secuencias sobre sus genes diana. La idea es comprender no sólo cómo se regula la activación de estos genes en condiciones fisiológicas, sino también cómo ciertas mutaciones en secuencias reguladoras resultan en patologías cerebrales. De hecho, aunque todavía estén poco caracterizadas, se sabe que mutaciones que afectan a secuencias reguladoras están en la base de muchas patologías congénitas y que las mutaciones en el genoma no codificante constituyen la mayor parte de la variabilidad genética existente en una población. Por ello, en los últimos años se ha acuñado el término “enhanceropatías” para referirse a las mutaciones de secuencias reguladoras relacionadas con condiciones patológicas. Además, se piensa que cambios en la regulación de los genes de desarrollo han contribuido de manera importante a la evolución de las especies y, en particular, a la de la corteza cerebral de humanos y primates.
Como modelo de estudio me centraré inicialmente en el gen TBR2, que juega un papel clave en el desarrollo de la corteza cerebral y cuyas mutaciones en regiones genómicas supuestamente reguladoras llevan a importantes malformaciones cerebrales. Además, cambios en la actividad de TBR2 han sido relacionados con la evolución de la corteza cerebral en mamíferos. Finalmente, la región genómica que contiene este gen presenta una compleja organización 3D, por lo que su estudio podría ayudar a comprender mejor los principios generales de la interrelación entre arquitectura 3D del ADN, actividad enhancer y regulación génica.
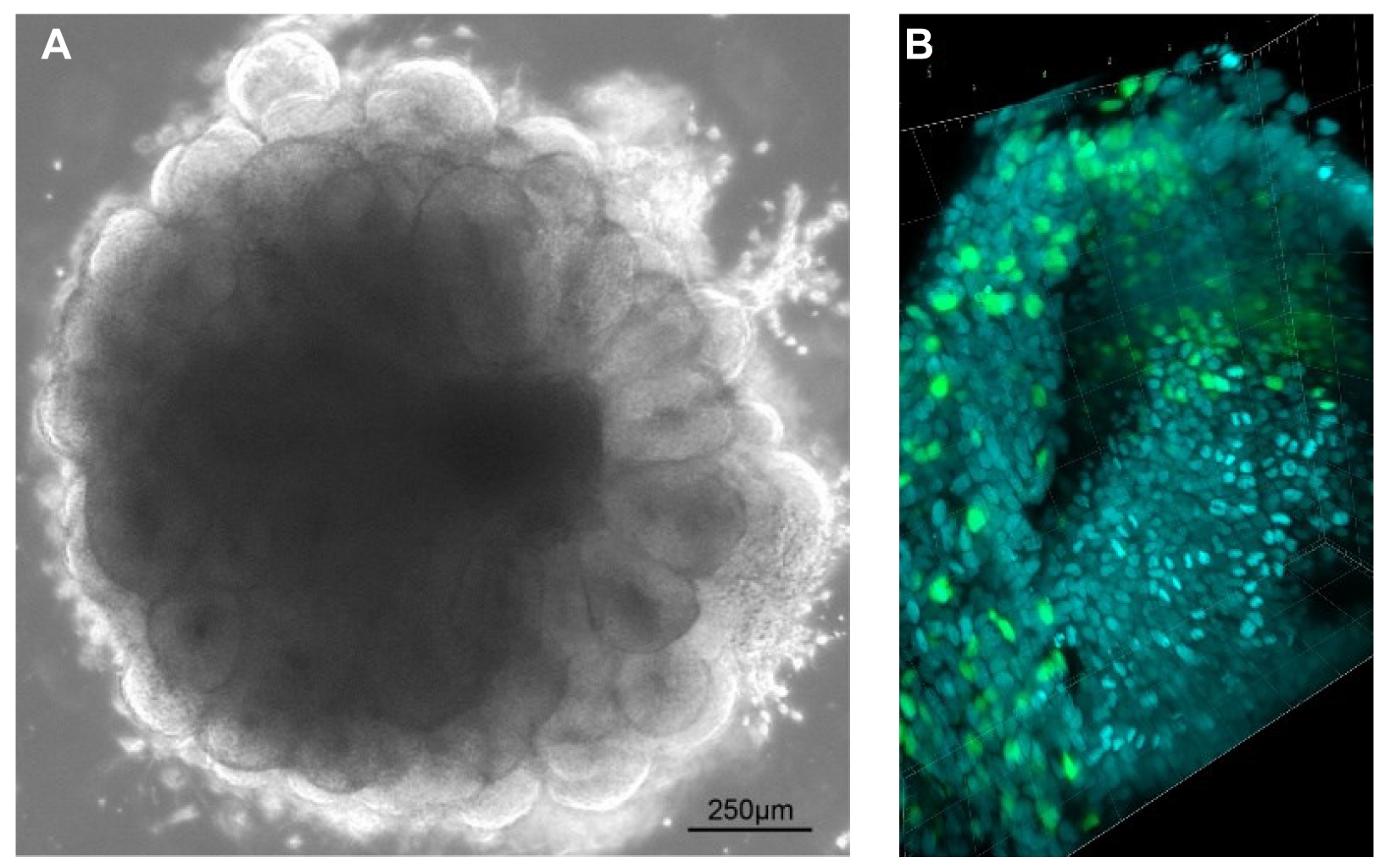
El estudio de secuencias reguladoras humanas, y del efecto de sus mutaciones, está limitado por la considerable divergencia evolutiva entre el genoma no codificante humano y el de otras especies comúnmente utilizadas como sistema modelo (como el ratón, el pollo o el pez cebra). Además, puesto que los genes de desarrollo suelen estar controlados por combinaciones de varios enhancers, su estudio suele requerir complejos experimentos bioquímicos y de edición genética, lo cual implica costes y tiempos experimentales elevados, así como el uso de un número importante de animales de experimentación. Para obviar estas limitaciones, mi proyecto se basa en la utilización de “mini-cerebros”: cultivos celulares tridimensionales derivados de células pluripotentes humanas que reproducen el desarrollo y la estructura de la corteza cerebral humana, y denominados organoides corticales (Figura 3).
Por tanto, mi proyecto se sitúa a caballo entre los dominios de la biología del desarrollo y de la genómica funcional/regulación génica, con vistas a poder relacionarse, en el futuro, con la investigación clínica de enfermedades cognitivas congénitas.
El programa Ramón y Cajal es un muy buen programa de atracción de talento investigador, con una clara finalidad de reclutar científicos que hemos realizado una experiencia profesional en el extranjero permitiendo la creación de nuevos equipos de investigación. El Ramón y Cajal ofrece un buen horizonte temporal y está bien coordinado con la convocatoria de Proyectos de Generación de Conocimiento para la obtención de los fondos necesarios para el proyecto de investigación. Además, ofrece la perspectiva de estabilización de los equipos emergentes.
En mi opinión, uno de los puntos a mejorar es que la convocatoria, muy dependiente de la consulta del BOE, hace complicado su acceso a candidatos extranjeros de habla no hispana y que desconocen el funcionamiento del sistema de investigación español. Esto, a mi entender, limita la atracción de candidatos que no hayan realizado al menos una etapa profesional en España. Además, el proceso de selección debería comportar una presentación más detallada del proyecto de investigación para poder evaluar mejor la viabilidad y el impacto de las propuestas, como un criterio importante para la selección de candidatos. Al mismo tiempo, creo que deberían establecerse dos posibles “itinerarios” profesionales, bien definidos en la convocatoria, que diferencien entre investigadores que aspiran a crear su propio equipo de investigación y los que prefieren trabajar en el contexto de otro equipo ya estructurado. Ambos perfiles deberían responder a requisitos y criterios de evaluación específicos y la forma para estabilizar a estas dos categorías debería también adaptarse consecuentemente.
Sin duda el centro de acogida de un candidato juega un papel importante en la realización del proyecto y en las perspectivas del(a) aspirante para poder establecer y evolucionar su equipo de investigación a medio/largo plazo. A mi parecer, dicha elección debe ponderarse detenidamente mediante entrevista con el centro de acogida antes de presentar la solicitud, aunque esto no sea un requerimiento estricto de la convocatoria.
En mi trayectoria profesional he tenido la enorme suerte de estar en contacto con científicos de enorme calidad profesional, intelectual y humana tanto en la supervisión de mis investigaciones como en el contexto de diferentes proyectos colaborativos.
Tanto mi directora de tesis, Paola Bovolenta, como mi supervisor posdoctoral, Denis Duboule, han impactado enormemente en mi desarrollo profesional por su apoyo científico y por su ejemplo de dedicación al trabajo, capacidad de análisis de las preguntas biológicas e integridad científica. De Paola he recibido, además, un enorme apoyo personal en los momentos difíciles y una ayuda enorme para la toma de decisiones clave en mi carrera, por lo que diría que ella es, sin duda, la persona que más ha impactado en mi desarrollo como científico.
Creo que la investigación biológica tiene mucho que aportar a la sociedad en las próximas décadas. En mi opinión su aporte puede (y debe) abarca dos aspectos diferentes: en cuanto fuente de progreso tecnológico y de conocimiento, por una parte, y como motor de progreso socio-cultural por otra.
En el primer aspecto, en las últimas décadas hemos presenciado un gran número de innovaciones y/o descubrimientos biológicos de gran impacto para el avance de la investigación biomédica/clínica y con una repercusión muy directa en la sociedad, como la secuenciación de genomas (incluido el genoma humano), el aislamiento y cultivo de células pluripotentes o las técnicas de edición genética. Por otro lado, creo que avances tecnológicos claves podrán venir de estudios de ingeniería biomimética: el estudio de diseños y estrategias de los seres vivos para adaptarse a su ambiente, como por ejemplo el diseño de materiales, la forma en la que explotar los recursos de forma eficiente y circular, generar y almacenar energía a temperatura ambiente gracias a reacciones químicas controladas, etc. En mi opinión, esta tendencia innovadora está destinada a mantenerse e incluso aumentar en las décadas futuras.
En el otro aspecto, coincido totalmente con los principios de crear una sociedad y una economía basada en el conocimiento. Los científicos, con ayuda de los políticos, debemos asumir el compromiso y el reto de difundir la ciencia a la sociedad y que su conocimiento sea considerado como un elemento esencial del bagaje cultural medio. Esto permitiría un mejor debate sobre temas relacionados con la ciencia de gran actualidad, como el uso de vacunas y antibióticos, el cambio climático, los organismos modificados genéticamente o el balance entre economía y explotación de los recursos naturales. Sin embrago, no me refiero solamente a la divulgación del conocimiento científico en sí, sino también al método por el cual la ciencia contrasta hipótesis y elabora conclusiones objetivas. Esto contrasta muchas veces con como la sociedad debate sobre cuestiones controvertidas y divisorias, donde prima la necesidad de convencer a una determinada audiencia más que la de llegar a un consenso sobre la interpretación de los hechos o datos.
En lo que se refiere a mi investigación, espero poder contribuir al avance del conocimiento sobre cómo la interpretación de la información contenida en el genoma permite el desarrollo correcto de un organismo y de cómo mutaciones en esta información genética (y en particular en las secuencias reguladoras) llevan a alteraciones del desarrollo cerebral. Esta investigación permitirá dar respuesta al origen de muchas patologías genéticas y, potencialmente, identificar estrategias para su diagnóstico temprano.

Leonardo Beccari nació en Italia en 1983 y estudió Biología en la Universidad de Málaga. Durante los dos últimos años de carrera se incorporó al Departamento de Biología celular, Genética y Fisiología animal, bajo la Supervisión de la Dra. María Dolores López Ávalos donde realizó su tesis de licenciatura. Posteriormente, se unió al laboratorio de la Prof. Paola Bovolenta, gracias a una beca de introducción de la investigación del CSIC (JAE-Intro) y, posteriormente, a un contrato pre doctoral de la Comunidad Autónoma de Madrid. Allí realizó su tesis doctoral, (que defendió en Mayo de 2011) centrándose en el estudio de las redes de regulación génica que controlan el desarrollo temprano del cerebro de vertebrados, utilizando como sistema modelo el pez Medaka. El trabajo de esos años permitió la publicación de dos artículos de investigación (en las revistas Development y Journal of Biological Chemistry) y una revisión como primer autor, así como de otras siete publicaciones como colaborador. En 2012 se incorporó al laboratorio del Prof Denis Duboule, en Ginebra, donde estudió cómo la interrelación entre de la organización 3D del genoma y la actividad secuencias de ADN reguladoras permite el control fino de la activación de los genes de desarrollo, centrándose en la regulación de los genes Hoxd de ratón, como modelo de estudio. Estos trabajos resultaron en la publicación de cuatro artículos de investigación como primer o coprimer autor (2 en la revista Genes and Development y uno en Nature), un artículo como último autor (Proceedings of the National Academy of Sciences-PNAS) y 5 artículos como colaborador. En 2020 se incorporó al Instituto NeuroMyoGene (Lion) para establecer su grupo de investigación, dedicado al estudio de los mecanismos de regulación génica que controlan el desarrollo de la corteza cerebral y cómo mutaciones que afectan a secuencias reguladoras conducen a defectos cerebrales congénitos. Tras haber sido seleccionado en la convocatoria del programa Ramón y Cajal (2020/21), planea incorporarse al Centro de Biología Molecular Severo Ochoa (Madrid) en Mayo 2022 para establecer su investigación en España


